即可将网页分享至朋友圈
无论是追踪未来流行病的蔓延,还是确定流行病爆发期间最佳分发疫苗的地区,今天的流行病研究人员都依赖于可靠的计算机模型。在流行病初期,经典的理论往往假设一个感染态节点平均新感染的人数(即基本再生数)几乎不变。这个假设成立吗?
近日,我校大数据研究中心博士生刘权辉和流行病传播界领军人物、美国东北大学杰出教授Alessandro Vespignani等人在《美国科学院院刊》发表研究论文。他们发现,“这一再生数在流行病初期是随时间变动的”。刘权辉为论文第一作者,电子科技大学为第一完成单位。
早期,由于无法收集到大规模真实的接触数据,常用简单的均匀网络来模拟流行病的传播,从而得到流行病初期感染人数随基本再生数呈指数增长。以最近非洲猪瘟为例,新引进的一头病猪感染了两头猪,这两头猪各自感染了另外两头猪,依次类推,从而得到病猪数目随时间增长的关系为1,2,4,8,16,32,64,128,……。当许多猪被疾病感染的时候,由于很难找到更多健康态的猪来感染,病猪数目随时间增长曲线将趋于稳定。
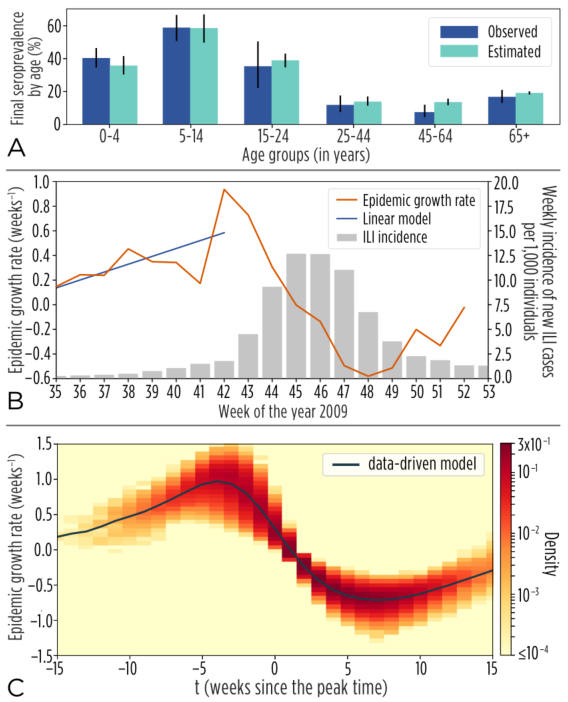
刘权辉等人的模型能够更精确描述真实流行病爆发的情况
刘权辉等人基于意大利和荷兰两个国家高度详细的社会人口统计数据,建立了包含家庭成员、公司同事、学校师生和社区任意两人间更现实的接触模型,发现真实网络上流行病并不是以简单的指数增长方式蔓延的。他们收集了有关2009年在意大利和荷兰两个国家爆发的猪流感数据,在建立的模型上模拟并检测该模型是否能够准确地与真实流行病的传播保持一致。他们发现,与经典定义的模型相比,他们构建的模型能够更准确地追踪和预测流行病的传播。刘权辉等人还发现,基于流行病爆发期间每日感染人数,利用机器学习方法可以较准地确定随时间变化的再生数。
该文在2018年11月被《美国科学院院刊》刊发,同时也被《美国科学院院刊》和美国东北大学重点报道。《美国科学院院刊》在综合性期刊中排名第三,仅次于《Nature》《Science》。该刊收录的文献涵盖生物、物理、社会科学和计算机科学,2017年影响因子为9.504。
论文链接:
http://www.pnas.org/content/early/2018/11/21/1811115115
编辑:李果 / 审核:王晓刚 / 发布:陈伟


