即可将网页分享至朋友圈
近日,电子科技大学生命科学与技术学院植物基因组工程研究团队与生物信息学研究团队共同在国际著名期刊《自然-通讯》(Nature Communications)发表了题为“Efficient plant genome engineering using a probiotic sourced CRISPR-Cas9 system”的研究论文。该研究针对植物基因组编辑工具开发瓶颈问题,聚焦国家重大战略需求,联合生物信息学分析方法,基于大数据挖掘,在细菌基因组中挖掘到了一个新型的CRISPR-Cas9系统LrCas9。

基因组编辑是在基因组水平上对特定DNA序列进行人工改造的遗传操作技术,是生命科学中的核心技术。目前,常用的基因组编辑系统是CRISPR-Cas9系统,开发此系统的两位科学家也因此获得2020年诺贝尔化学奖。在目前所开发的CRISPR-Cas9系统中,来源于病原菌化脓性链球菌(Streptococcus pyogenes)的SpCas9系统应用最为广泛。但是其致病菌来源的属性可能使得公众对生物安全充满担忧。更重要的是,相关技术专利为外方掌握,基因组编辑底层技术仍存在“卡脖子”的情况。
LrCas9来源于非致病菌鼠李糖乳杆菌(Lactobacillus rhamnosus GG),经后续实验发现LrCas9系统特异识别5’-NGAAA-3’ PAM位点,在水稻、小麦、番茄及落叶松中均可实现有效的基因组编辑。同时,LrCas9在相同的位点较LbCas12a及SpCas9变体(SpCas9-NG、SpRY)均展现了更高的编辑活性。结合LrCas9系统识别A/T富集PAM的特性,在水稻T0代植株中实现了针对OsWx基因的启动子编辑,获得了直链淀粉含量变化的新种质。经spacer 1bp mismatch、GUIDE-seq分析及CRISPR-GE分析,证明了LrCas9的高特异性。此外,本文还基于LrCas9开发了碱基编辑工具及基因表达调控工具,丰富了LrCas9编辑工具箱。
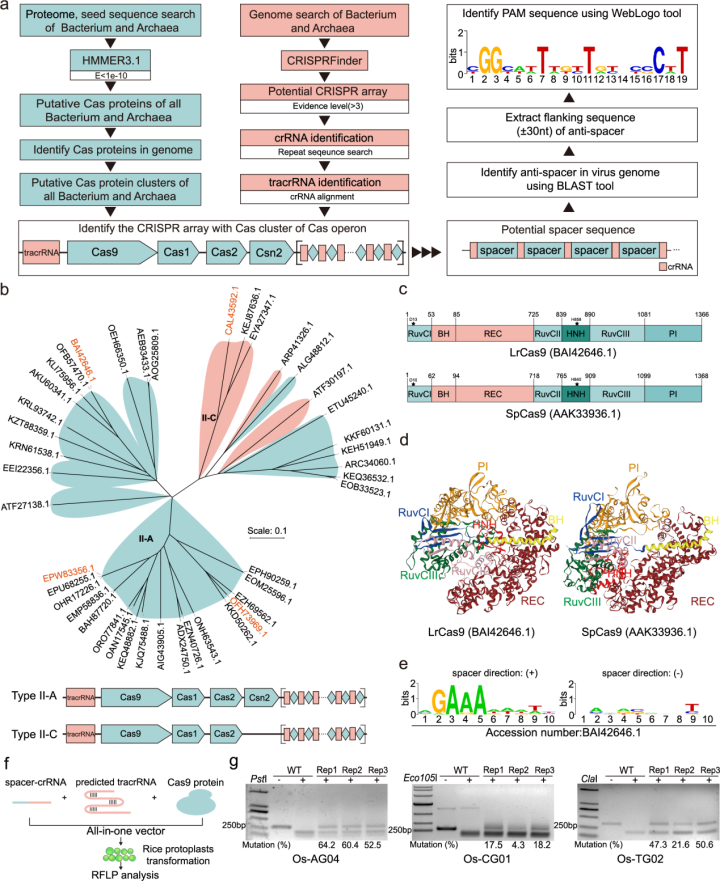
该工作契合了生命学院生物研究与电子信息融合的发展特色,体现了学院“立足生命科学与电子信息领域交叉融合”的特色理念,为进一步推动并实现生命学院多学科交叉提供了良好借鉴。此研究所开发的LrCas9系统已经申请并获批中国知识产权,是具有自主知识产权的新型基因组编辑系统,且其高效、高特异性的优良编辑特性丰富了现有基因组编辑工具库。值得一提的是,该论文发表后被杂志编辑部标记为亮点研究重点向读者推荐。
经生命学院生物技术专业本、硕、博培养的博士后仲昭辉博士和已毕业硕士生唐钟杰、向舒月为论文共同第一作者。张勇教授、黄健教授为共同通讯作者。电子科技大学生命科学与技术学院为第一作者单位和通讯单位。Nature Communications是高水平综合性的国际学术期刊,2023年最新影响因子为16.6。
编辑:张闻起 / 审核:李果 / 发布:陈伟


